ภารระงานที่ 4
3 การจำลองของโครงสร้างของ DNA
การจำลองตัวเองของดีเอ็นเอ (DNA replication) เป็นกระบวนการทางชีววิทยาที่เกิดขึ้นในสิ่งมีชีวิตทุกชนิดเพื่อจำลองดีเอ็นเอของตนเอง
กระบวนการนี้เริ่มจากดีเอ็นเอสายเดี่ยวสร้างดีเอ็นเออีกสายที่เป็นคู่สมของตนจนกลายเป็นดีเอ็นเอเกลียวคู่
กระบวนการเป็นแบบกึ่งอนุรักษ์ (semiconservative
replication) มีการตรวจสอบความถูกต้องของกระบวนการเพื่อป้องกัน
ภาพที่1
แสดงการจำลองตัวเองของ DNA
รูปแบบการจำลองของตัวเองของดีเอ็นเอในสิ่งมีชีวิต
แบบซีตา (Theta
model) พบในสิ่งมีชีวิตที่มี DNA เป็นวงกลม
เช่นแบคทีเรีย โดยช่วงหนึ่งดีเอ็นเอจะมีรูปร่างคล้าย θ การจำลองจะเริ่มที่จุดๆหนึ่งซึ่งจะโป่งออกเป็นห่วงโดยมี Replication
fork อยู่ที่ปลายแต่ละด้านของจุดเริ่มต้น เกิด 2 ทิศทาง สุดท้ายจะได้ดีเอ็นเอ 2 วง
แบบซิกมา (Sigma
model) ช่วงหนึ่งดีเอ็นเอมีรูปร่างคล้าย ς พบใน DNA ที่เป็นวงกลม ที่จุดเริ่มต้นจะเกิดช่องขาด
ขึ้นเนื่องจากพันธะฟอสโฟไดเอสเทอร์ถูกตัด ดีเอ็นเอสายหนึ่งยืดยาวออกไปเป็นแม่แบบ
เมื่อสิ้นสุดจะได้ดีเอ็นเอเกลียวคู่เป็นวง 1
วง และเป็นเส้น 1 เส้น
แบบวาย (Y-shaped
model) เป็นแบบที่พบมากที่สุด โดยจุดเริ่มต้นมีมากกว่า 1 จุด แต่ละจุดโป่งออกเป็นห่วงกระจายทั่วไป เกิด 2 ทิศทางพร้อมกัน
ในที่สุดแต่ละห่วงจะมาชนกันได้ดีเอ็นเอใหม่ 2 สาย
การจำลองตัวเองของ DNA
ภาพที่2
แสดงรหัสพันธุกรรม
ภาพที่ 3
ความแตกต่างระหว่างเซลล์โปรคาริโอตและเซลล์ยูคาริโอต
การเริ่มต้น (Initiation) ในดีเอ็นเอของแบคทีเรียนั้นจะมีจุดสำหรับเริ่มจำลองดีเอ็นเอ
มีโปรตีนเข้ามากระตุ้นให้ดีเอ็นเอที่จุดเริ่มต้นดังกล่าวคลายตัว เฮลิเคสเข้ามาตัดพันธะไฮโดรเจนระหว่างสายดีเอ็นเอ ดีเอ็นเอบายดิงโปรตีนมาจับเพื่อป้องกันไม่ให้สร้างพันธะไฮโดรเจนต่ออีก
การสร้างดีเอ็นเอสายใหม่ (Elongation) เมื่อดีเอ็นเอทั้งสองสายแยกจากกันแล้ว DNA polymerase III จะเข้ามาตรงจุดแยกเพื่อสร้างดีเอ็นเอสายใหม่
เนื่องจาก DNA pol III มีคุณสมบัติในการสร้างดีเอ็นเอสายใหม่จาก
5′
ไป 3′
เท่านั้น ซึ่งต้องการแม่แบบที่เป็นสาย 3′ ไป 5′ แต่ดีเอ็นเอแม่แบบมีทั้งที่เป็น 3′ ไป 5′ และ 5′ ไป 3′ ดังนั้น
การสร้างสายดีเอ็นเอจึงแบ่งเป็น 2 แบบดังนี้
สายต่อเนื่อง (Leading stand) คือสายที่เป็น 3′ ไป 5′ ในสายนี้ DNA polymerase
III จะสร้างดีเอ็นเอสายใหม่ได้อย่างต่อเนื่อง เริ่มต้นโดย Primase
สร้างไพรเมอร์ที่เป็นอาร์เอ็นเอสายสั้นๆ ขนาด 10 – 26 นิวคลีโอไทด์ เข้ามาจับกับดีเอ็นเอตรงจุดแยก จากนั้น DNA
polymerase III จะเติม dNTPs เข้ามาในทิศทาง 5′ ไป 3′ ไปเรื่อยๆ
สายไม่ต่อเนื่อง (Lagging stand) เนื่องจากสายนี้ดีเอ็นเอแม่แบบเป็น
5′
ไป 3′
การสร้างดีเอ็นเอเป็นสายยาวไปทีเดียวจึงเกิดขึ้นไม่ได้
แต่จะใช้วิธีให้สายดีเอ็นเอโค้งงอผ่าน DNA pol III เพื่อให้ DNA
polymerase III สร้างดีเอ็นเอสายใหม่ในทิศทาง 5′ ไป 3′ เป็นชิ้นเล็กๆ เรียกชิ้นเล็กๆนี้ว่าชิ้นส่วนโอคาซากิ
(Okazaki fragment) โดย Primase สร้างไพรเมอร์ที่เป็นอาร์เอ็นเอสายสั้นๆ
สำหรับการสร้างชิ้นส่วนโอคาซากิแต่ละชิ้น หน่วยย่อยเบตาของ DNA polymerase
III จะเข้ามาจับที่ไพรเมอร์และเชื่อมต่อกับ DNA polymerase
III เมื่อหมดชิ้นจะสร้างชิ้นใหม่ หน่วยย่อยเบตาอันเดิมจะหลุดไป
หน่วยย่อยเบตาอันใหม่จะเข้ามาจับกับไพรเมอร์อันต่อไป เป็นเช่นนี้ไปเรื่อยๆ
การตรวจสอบ
ส่วนของไพรเมอร์ที่เป็นอาร์เอ็นเอจะถูกตรวจสอบด้วย DNA polymerase I และสร้างดีเอ็นเอซ่อมแซมส่วนที่ตัดออกไป
จุดขาดที่เกิดขึ้นบนสายดีเอ็นเอ เนื่องจาก DNA polymerase I ไม่ได้ต่อพันธะฟอสโฟไดเอสเทอร์ตรงปลาย
5′
ของจุดตัดจะถูกเชื่อมด้วย ดีเอ็นเอไลเกส โดยสายต่อเนื่องจะตัดครั้งเดียว
ส่วนสายไม่ต่อเนื่องจะตัดไพรเมอร์ของชิ้นส่วนโอคาซากิทุกชิ้น
การสิ้นสุด (Termination)
ระหว่างจุดเริ่มต้นแต่ละแห่งจะมีจุดสิ้นสุดของเรพลิเคชันอยู่ด้วย
มีขนาด 20 คู่เบส เรียกว่า ter sequence ซึ่งจะมีโปรตีนที่จดจำตำแหน่งนี้เข้ามาจับเพื่อบอกให้ DNA
polymerase III รู้ว่าจำลองดีเอ็นเอมาครบรอบแล้ว
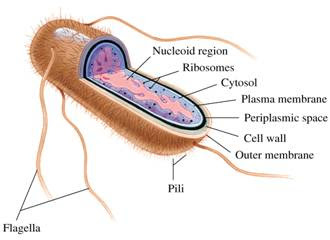
ภาพที่ 4
เซลล์โพรคาริโอต
การจำลองตัวเองในยูคาริโอตมีความซับซ้อนกว่า
โดยจุดเริ่มต้นนั้นจะมีโปรตีนที่จดจำตำแหน่งนี้โดยเฉพาะเข้ามากระตุ้นให้ดีเอ็นเอคลายตัว
DNA polymerase มีหลายชนิด
โดยในนิวเคลียสใช้ DNA polymerase α และ DNA
polymerase δ โดย DNA polymerase α ทำหน้าที่คล้าย DNA polymerase III ในโปรคาริโอต DNA polymerase δ ทำหน้าที่คล้ายหน่วยเบตาของ
DNA polymerase III และ DNA polymerase ε ทำหน้าที่คล้าย DNA polymerase I ในโปรคาริโอต การสิ้นสุดเรพลิเคชันในยูคาริโอต
เกี่ยวข้องกับการสังเคราะห์โครงสร้างพิเศษที่เรียกเทโลเมียร์ที่ตอนปลายของโครโมโซม
คำถาม
1 รูปแบบการจำลองตัวเองของดีเอ็นเอในสิ่งมีชีวิต
มีอะไรบ้าง
ตอบ
2 การจำลองของตัวเองของโปรคาริโอตคืออะไร
2 การจำลองของตัวเองของโปรคาริโอตคืออะไร
ตอบ
3.การจำลองของตัวเองของยูคาริโอตคืออะไร
ตอบ